Javier Carrera es un científico-ingeniero español especializado en biología sintética. Trabaja en el diseño de modelos computacionales que simulan la compleja biología de los microrganismos.
La revista Science publica hoy un artículo suyo en el que anuncia la creación del primer motor tecnológico capaz de integrar y simular todo el conocimiento acumulado durante décadas en Escherichia coli, la bacteria mejor estudiada en biológica molecular.
Este desarrollo, que incorpora bases similares de software a las usadas en Google Maps, tendrá profundas implicaciones en medicina proactiva y biotecnológica. En 2017 esta innovación le mereció a Javier Carrera la nominación al Innovator Under 35, la lista anual que reconoce a innovadores sobresalientes menores de 35 años elaborada por la MIT Technology Review.
¿En qué consiste la base tecnológica del primer motor capaz de integrar y simular todo el conocimiento acumulado durante décadas en Escherichia coli?
Hemos construido un modelo computacional que integra la mayor parte de los mecanismos biológicos conocidos hoy en día (que han sido proporcionados gracias a varias décadas de experimentos en la biología molecular de E. coli) mediante modelos matemáticos. Estos mecanismos biológicos son las pequeñas piezas del puzzle que representan el funcionamiento y modo de operación de E. coli dependiendo de las condiciones ambientales en las que vive: como ejemplos, señalaría nuestro microbioma, o simplemente medios de cultivo estándares usados en laboratorio.
Mediante esta tecnología, primero simulamos cómo cada gen de la bacteria induce ciertas funciones biológicas en ciertos momentos del ciclo celular; en segundo lugar, podemos cuantificar todos los componentes intra-moleculares en tiempo real (ADN, RNA, proteínas, moléculas derivadas del metabolismo, maquinaria celular como ribosomas o polimerasas – entre otros); y en tercer lugar podemos observar cómo ellos interactúan de una manera sincronizada. Todo ello con la finalidad de capacitar a los bio-ingenieros para optimizar y entender la biología de los microorganismos relacionados con la medicina o la biotecnología industrial.
¿Cómo se llega a un resultado así en tu carrera profesional?
Estudié ingeniería mecánica en España/Suecia (2006). Seguidamente, mis estudios de tesis fueron entre Valencia y Paris (2012), focalizados en el área de biología sintética (en el nexo de ingeniería y biología molecular), con el objetivo de diseñar modelos computacionales que pudieran servir para entender y simular la compleja biología que gobierna el funcionamiento de los microrganismos. Durante esta etapa tuve la oportunidad de trabajar con pioneros del campo en el MIT, lo que despertó mi interés por aterrizar en el sistema académico americano. Mi faceta en Stanford (2017) me ayudó a consolidar trabajos arrancados durante mi tesis, en colaboración con un equipo de ingenieros, biólogos moleculares, y científicos. Fruto de esa colaboración emergió lo que a día de hoy llamamos “whole-cell model”, el primer motor tecnológico capaz de integrar y simular todo el conocimiento acumulado durante décadas en Escherichia coli, la bacteria mejor estudiada en biológica molecular, con profundas implicaciones hacia una medicina proactiva y una biotecnología basada en ciclos rápidos de Design-Build-Test-Learn. Ese avance tecnológico apoyó mi nominación en 2017 al “Innovator Under 35”, otorgado por la MIT Technology Review. Finalmente, esta tecnología ha sido seleccionada por Science para describir casi una década de investigación en Stanford, respaldada en ejecución por uno de los primeros ingenieros clave de Google Maps.
¿Qué relación tiene vuestro desarrollo con la tecnología de Google Maps?
Debo aclarar que no hemos usado ninguna tecnología de Google. En concreto, Jerry Morrison (incluido en la lista de autores) se unió al proyecto en 2015. Es uno de los ex-ingenieros pioneros de Google Maps que, incentivado por este proyecto ambicioso, se reincorporó al mundo académico y se unió a nuestro equipo en Stanford. Como experto en desarrollar tecnologías con impacto a millones de usuarios (como Maps), Morrison incorporó grandes dosis de buenas prácticas en ingeniería de software para generar una arquitectura computacional que ayudara a lanzar simulaciones que son masivas en cuanto a los recursos (computacionales) necesarios para ser ejecutadas.
Este desarrollo tecnológico, ¿puede facilitar una nueva generación de antibióticos mediante computación? ¿Impactará también otras ingenierías?
Uno de los principales objetivos de esta tecnología ha sido facilitar la simulación del funcionamiento de antibióticos para mejorar su eficiencia y luchar contra bacterias patogénicas con resistencia a los antibióticos clásicos. Esta tecnología abre las puertas a poder diseñar antibióticos más efectivos suplementándolos con moléculas que incentivan su eficiencia, e incluso explorando combinaciones de antibióticos actualmente presentes en mercado.
Hay que tener en cuenta al respecto que el mercado de los antibióticos no ha sido sostenible y que ha derivado en un colapso masivo durante los últimos tiempos en los departamentos de antimicrobiales de las grandes farmacéuticas (por ejemplo, Novartis-Emeryville en 2018). Es un mercado con incentivos débiles: costes elevados para producir nuevos antibióticos, fundamentalmente debido a fases de desarrollo y ensayos clínicos que sobrepasan los 10 años en muchos casos. Además, generan beneficios muy reducidos cuando nuevos fármacos son sacados a fases comerciales. Nuevas tecnologías para acelerar el proceso efectivo de R&D son necesarios con máxima urgencia en este sector: se estima que en las próximas 2-3 décadas, la tasa de mortalidad asociada a infecciones bacterianas superará a la que asociamos a enfermedades puramente relacionadas con cáncer, debido a que las bacterias están adquiriendo resistencia los antibióticos actuales. Los gobiernos y las industrias farmacéuticas no han activado planes para abordar este gran problema de la resistencia a antibióticos.
Respecto a otras disciplinas científico-tecnológicas plenamente consolidadas (como la ingeniería mecánica o electrónica), se necesitan plataformas de simulaciones que capaciten a los bioingenieros el modelado de cómo funcionarían nuevas terapias, sin necesidad inmediata de ejecutar experimentos (con costes elevados) para seleccionar las terapias “más prometedoras». Esta tecnología publicada ahora en Science activa el paradigma de experimentación en biología molecular guiado por simulaciones computacionales, abriendo puertas a una nueva era en la que la biología de sistemas ya acaba de demostrar que facilita diseños más efectivos.
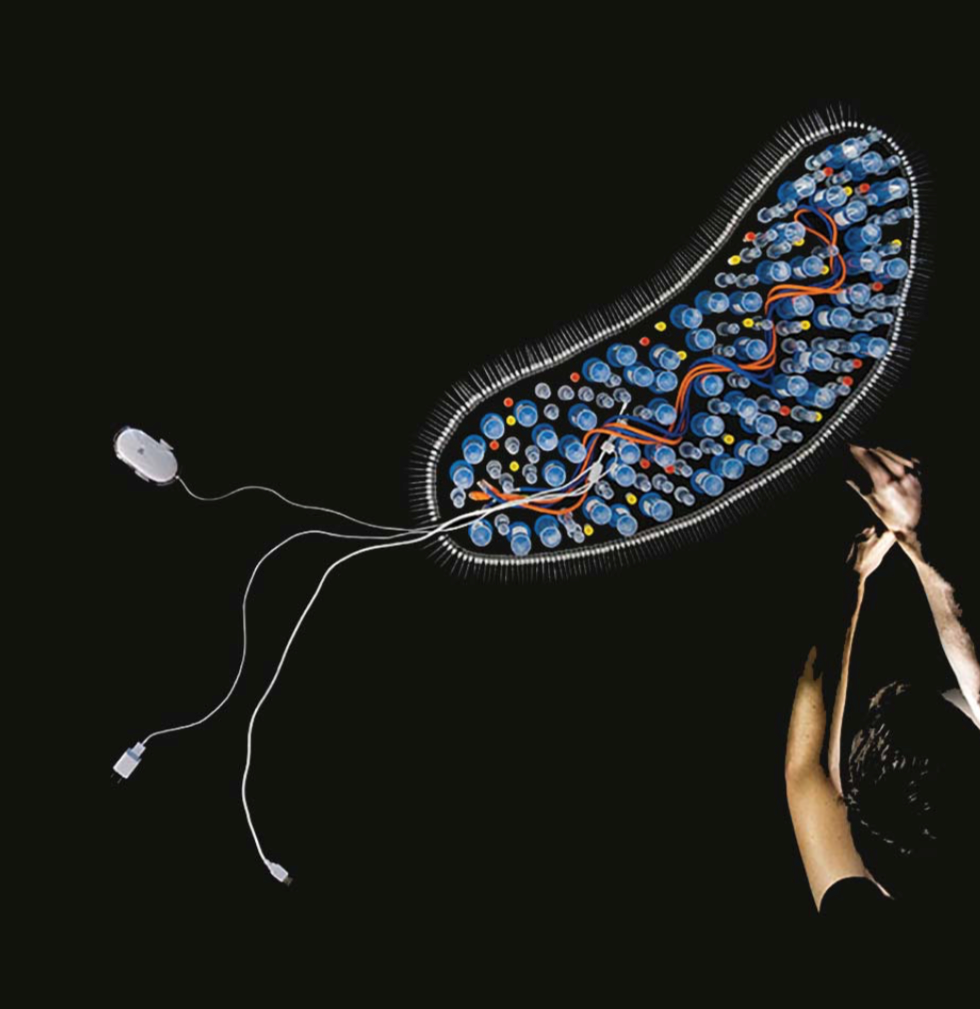
¿Cómo una plataforma de integración de conocimiento y simulación puede acelerar el descubrimiento de fármacos efectivos para luchar contra la actual pandemia?
Es de relevancia resaltar la cercanía entre los problemas que experimentamos actualmente para encontrar vacunas/fármacos efectivos contra COVID-19 y esta nueva metodología para simular biología y acelerar descubrimientos. Actualmente, estamos experimentando una generación masiva de datos con relación a SARS-CoV-2 para aprender rápidamente cómo este virus patogénico interactúa y progresa con seres humanos a distintas escalas (en tiempo y a nivel molecular). Es uno de esos momentos críticos en la historia de la medicina, en el que deberíamos reconocer que es clave centralizar y coordinar la interrogación científico-experimental (como Francis Crick, premio Nobel al descubrimiento estructural del ADN, ya defendió en uno de sus monográficos hace cuatro década) y posterior agregación de datos/conocimiento para luchar contra esta enfermedad que nos ha traído consecuencias sin precedentes. En este contexto, esta tecnología ha sido pionera para integrar todo el conocimiento generado en la biologia de E. coli durante más de cuatro décadas, a lo largo de centenares de laboratorios en el mundo, millones de experimentos, y billones de datos, con el objetivo de identificar cuál sería el siguiente experimento que nos capacitaría para descubrir otro pequeño rincón desconocido del funcionamiento complejo de sistemas biológicos, algo que es indiscutiblemente necesario en tiempos de pandemia para traer fármacos a la mesa lo antes posible. Todo segundo cuenta, todo experimento ha de ser para descubrir algo nuevo, y nuevos datos adquiridos deben solo aportar más piezas del puzzle de una manera estratégica y coordinada.
¿Cómo ha sido el proceso que ha convertido este desarrollo tecnológico en un éxito también empresarial?
Es hacia mitad del 2012 cuando personalmente descubro que un laboratorio de Stanford está desarrollando una tecnología emergente con la que yo había soñado durante mis estudios de doctorado (2007-2012): ordenadores capaces de simular toda la biología que se conoce de una bacteria. Considero sustanciales los avances que logramos en aquellos tiempos de tesis, pero mis avances de entonces distaban mucho de ser un motor útil en el mundo no-académico. Finalmente, me uno a este proyecto en 2013, aterrizando en uno de los ecosistemas más innovadores, Silicon Valley. Ya unido al proyecto, empezamos a observar que los tiempos de desarrollo tecnológico superan sustancialmente los plazos que habíamos programado. En 2015 decido aparcar por unos meses el desarrollo tecnológico (continuado por el resto del equipo) y acercarme al mundo de la industria mediante un “short MBA” en la escuela de negocios de Stanford. En retrospectiva, pienso que fue clave poder inyectar dosis de “customer discovery” para girar ligeramente el enfoque tecnológico-científico inicialmente diseñado, ofrecido únicamente a laboratorios puramente académicos. Hoy en día, ya hablamos de algo con un impacto mucho mayor: grandes tecnológicas de Silicon Valley ya han negociado con la Universidad la adquisición de la licencia que respalda este nuevo motor tecnológico para uso no-académico. Finalmente, remarcaría el hecho de que este motor tecnológico no necesariamente hubiera escalado más rápido con recursos financieros en sus inicios. Más bien, considero que es el resultado de una sólida alineación entre incentivos dictados por mercados en biotecnología/medicina explotados eficientemente por líderes en ingeniería y biología molecular (mis mentores en Stanford). Esa sí que resulta una de las grandes limitaciones impuestas fuera del ecosistema único que ofrece Silicon Valley.
Referencia
Simultaneous cross-evaluation of heterogeneous E. coli datasets via mechanistic simulation. Javier Carrera et al. Science 24 Jul. 2020:Vol. 369, Issue 6502, eaav3751. DOI: 10.1126/science.aav3751














Siempre se ha pensado que la nueva tecnología solo sirve para los ejércitos y los gobiernos, esto demuestra que en la rama de la ciencia es más que importante.