
La bioinformática, una disciplina relativamente joven, permite identificar genes, entre los aproximadamente 23.000 que componen el genoma humano, que están involucrados o son responsables de una determinada enfermedad.
Juan Manuel Luque, biólogo y bioinformático de la Unidad de Biología Computacional, de la Unidad Funcional de Investigación de Enfermedades Crónicas del Instituto de Salud Carlos III (Madrid), colabora en un Proyecto Internacional con el Institute of Population and Health College of Life Science (Northwest University, en Xian, China), titulado Análisis de genes relacionados con la Discapacidad Intelectual No Sindrómica (NS-ID) en una población china, usando secuenciación masiva (NGS) y métodos bioinformáticos.
La discapacidad intelectual (ID en sus siglas en inglés), hasta hace muy poco tiempo referida como Retraso Mental, no es una entidad única de enfermedad, sino un grupo de una gran y heterogénea colección de desórdenes sindrómicos y no sindrómicos, con las capacidades intelectuales disminuidas como marca o denominador común.
Los pacientes con ID sindrómico (S-ID) tienen, además de las capacidades intelectuales disminuidas, otras anomalías adicionales como rasgos dismórficos o defectos metabólicos. En cambio, los pacientes con ID no sindrómico (NS-ID) sólo muestran clínicamente una disminución de las capacidades intelectuales. Sin embargo, esta división entre ambos tipos de ID a menudo es arbitraria y no está tan clara. La ID puede ser causada por defectos genéticos,así como por efecto del medio ambiente.
“Mi labor”, explica el científico español, “consiste en analizar por técnicas y métodos bioinformáticos los archivos informáticos que contienen las secuencias de bases del genoma de cada paciente afectado de la enfermedad NS-ID, buscando los genes y procesos celulares alterados e implicados en la enfermedad.”
Utilizando un protocolo o procedimiento de análisis con tres programas bioinformáticos -SeqMule, wAnnovar y DAVID- se pueden identificar (a partir de los archivos de secuenciación de los pacientes) una lista de 386 genes del genoma humano implicados o responsables de la enfermedad NS-ID en una familia china, así como deducir un posible mecanismo molecular de la misma.
El proyecto comenzó en 2013 con el estudio de una familia china afectada de NS-ID a lo largo de tres generaciones (hijos, padres y abuelos) y la secuenciación masiva (NGS: Next Generation Sequencing) de siete miembros de la misma. Sus exomas (parte del genoma que codifica proteínas) fue analizado mediante métodos bioinformáticos y los programas mencionados.
Seis de los siete miembros secuenciados padecen la enfermedad y un miembro está sano y hace el papel de control.
Con el software SeqMule obtuvieron el primer archivo en formato VCF) de variantes génicas o mutaciones puntuales del exoma de cada paciente, a partir de los archivos de secuenciación NGS que salen del secuenciador y que se introducen en SeqMule.Se suelen obtener aproximadamente de 25.000-300.00 variantes génicas o mutaciones puntuales en cada archivo VCF de cada paciente.
Con wAnnovar obtuvieron la lista de variantes génicas filtradas frente a diversas bases de datos de variantes del genoma humano (seleccionando sólo las variantes presumiblemente causantes de NS-ID y descartando SNPs : polimorfismos de una base no patogénicos y otras variantes no relacionadas con la enfermedad), y obtuvieron la lista de genes candidatos causales de NS-ID para el exoma de cada paciente, a partir del archivo VCF procedente de SeqMule.
Se suelen obtener aproximadamente entre 400-500 variantes génicas responsables de NS-ID, así como los respectivos 400-500 genes candidatos de NS-ID que portan las variantes génicas patogénicas para el exoma de cada paciente.
Finalmente, con el software DAVID se obtiene un listado de las funciones de los genes candidatos de NS-ID a partir de la lista de genes obtenida en el paso anterior, así como un listado de los procesos celulares y pathways (vías de señalización molecular) en los que están implicados los genes candidatos de la lista. Con esta información, explica Luque, se puede deducir un posible mecanismo molecular de la enfermedad NS-ID.
Genes alterados
Analizados y comparados los genes alterados en cada uno de los siete miembros de la familia, y una vez descartados los genes comunes de la persona sana, queda una lista de 368 genes alterados candidatos comunes a dos o más personas secuenciadas.
“Eso implica que existe una clara base genética como causa de la NS-ID en esta familia”, afirma Luque. “La media de genes involucrados en una determinada enfermedad o patología está en torno a 400 genes, lo que se aproxima bastante a nuestro caso”.
“Muchos de los genes candidatos alterados que hemos encontrado coinciden en gran medida o son análogos con los referidos en la bibliografía científica, y hay otros que no figuran en esa bibliografía. Y lo mismo podemos decir respecto a los procesos celulares y pathways encontrados”, sigue. “Por tanto, podríamos estar ante nuevos hallazgos sobre las alteraciones, la etiología [causas] y el mecanismo molecular de la NS-ID”.
Tipos
Entre los genes alterados se observan varios tipos:
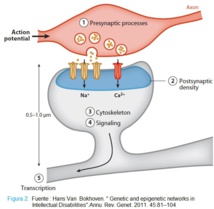
-Genes y sus proteínas implicadas en señalización celular y activación transcripcional a través del pathway Rho-GTPasas: sugiere que eventos como el tráfico de vesículas de neurotransmisores en espinas dendríticas y sinapsis, así como la organización del citoesqueleto son procesos importantes en el desarrollo del conocimiento normal y el aprendizaje del individuo, y si estos fallan provocan discapacidad intelectual (ID).
-Genes implicados en tráfico y liberación de neurotransmisores.
-Genes implicados en el remodelado de la cromatina: un mecanismo esencial para la expresión génica y el mantenimiento de la estructura de la cromatina, y también parece jugar un papel en la etiología de la enfermedad NS-ID, explica Luque.
-Genes implicados en el proceso de extensión o guía de axones (axon guidance). Este proceso forma redes nerviosas, circuitos neuronales o interconexión de neuronas. Las mutaciones o alteraciones en los genes y proteínas de este proceso “provocarían déficit o fallos en el desarrollo neuronal, en el aprendizaje y la memoria, dando lugar a NS-ID.
De nuevo son muchos genes y proteínas del citoesqueleto las implicadas en este proce-
so”.
-Genes implicados en el metabolismo de lípidos, fundamental para el correcto desarrollo del sistema nervioso. Es un tejido muy rico en lípidos y alteraciones en los genes y proteínas de este metabolismo contribuyen a la enfermedad NS-ID.
Aplicaciones
Entre las posibles futuras aplicaciones de los conocimientos y hallazgos obtenidos en este proyecto después del análisis bioinformático, y una vez validados experimental-
mente los mismos, estarían:
-El posible diagnóstico molecular de la NS-ID, usando un panel de genes alterados como biomarcadores.
-Una posible terapia, con el desarrollo de compuestos que actúen en las vías de señaliza-
ción/ pathways alteradas y que anulen o neutralicen esas alteraciones.
-Una posible edición o corrección de las alteraciones/mutaciones génicas identificadas, recurriendo a nuevas y prometedoras técnicas o métodos como el sistema de edición génica CRISPR /Cas 9.
-Lo más importante, según Luque: “Resaltar el gran potencial de este procedimiento secuencial con los tres softwares bioinformáticos, que se podría aplicar al estudio a nivel molecular de cualquier enfermedad, obteniendo así una información muy útil y valiosa de la misma”.
Referencia bibliográfica:
Hans Van Bokhoven: Genetic and epigenetic networks in Intellectual Disabilities. Annual Review of Genetics (2011). DOI: 10.1146/annurev-genet-110410-132512
Datos sobre el autor
Juan Manuel Luque Sánchez es Licenciado en CC. Biológicas y Máster en Bioinformática. Instituto de Salud Carlos III (Unidad Biología Computacional-UFIEC).

















Hacer un comentario